¡Sorpresa: No todo el ARN se transcribe a proteína!
Ya hablamos de que no todo el ADN era codificante, ahora sabemos que hay millares de fragmentos de ARN que no dan lugar a ninguna proteína. Son los denominados “ARN no codificantes” (ncRNA, en inglés), y los hay de muchos tipos. Ya entre los años 60 y 80 se descubrieron los ARN de transferencia (tRNA) y los ribosomales (rRNA); pero con el advenimiento de las nuevas tecnologías de secuenciación a gran escala (secuenciación masiva, o NGS por su sigla en inglés) se ha revelado que, sorprendentemente, la proporción de genoma transcripto a ARN pero que no produce proteínas es mucho mayor de lo que previamente se estimaba.
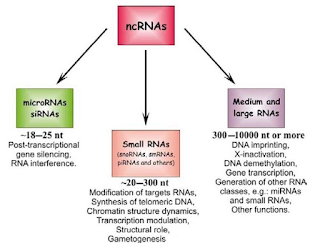
Estos transcriptos no codificantes son normalmente clasificados de acuerdo a su longitud, en ARNs no codificantes cortos y largos. Ejemplos de ARN no codificantes cortos (sncRNA) incluyen ARNs ribosomales (rRNAs), micro ARNs (miRNA), ARNs de interferencia o silenciamiento (siRNAs), y ARNs de interacción con proteínas PIWI (piRNAs). Estos pequeños ARNs suelen tener funciones regulatorias y están involucrados en procesos de silenciamiento o apagado de genes1.
Por su parte, los ARNs no codificantes largos (lncRNAs por su nombre en inglés, long noncoding RNAs) son definidos por oposición a los cortos, como aquellos transcriptos de ARN que presentan al menos 200 nucleótidos de longitud. Si bien el estudio de los lncRNAs ha sido históricamente relegado, en los últimos tiempos se ha comenzado a prestar particular atención a este subgrupo de ARNs, vinculándolos con variados procesos biológicos como diferenciación, proliferación y migración celular, apoptosis, respuesta inmune, desarrollo tisular, etc. Investigaciones recientes han mostrado que al menos algunos lncRNAs están asociados a procesos moleculares como regulación de la expresión de genes (a nivel de la transcripción, posttranscripcional, control de actividad de proteínas), modificaciones en la cromatina, organización de dominios nucleares, corte y empalme de ARNs mensajeros (splicing, en inglés), entre otros. Más aún, la alteración en la expresión de algunos lncRNAs se ha relacionado con la generación de tumores; incluso podrían actuar como oncogenes2.
¿Y cuál es el papel de estos ncRNA en el imprinting? Se ha demostrado que todos las regiones de impronta bien caracterizadas contienen al menos uno o dos lncRNA con un patrón de expresión inverso a los genes improntados3. Estos lncRNAs se localizan en las regiones diferencialmente metiladas o DMRs, que, como recordaréis son las regiones cuyas alteraciones daban lugar a las distintas enfermedades de impronta. De hecho, según su ubicación, los lncRNA se pueden clasificar en lncRNA intergénicos, lncRNA antisentido, lncRNA intrónicos y lncRNA potenciadores. Con la excepción de los lncRNA intrónicos, todos los lncRNA restantes se han implicado funcionalmente en la expresión específica del gen en función de su origen parental4. Los lncRNA pueden regular la expresión de ese gen complementario específico, ya sea a través del remodelamiento de la cromatina, el procesamiento alternativo de los mRNA (splicing) o la generación de siRNA. Estos últimos, junto a los miRNA representan mecanismos epigenéticos basados en ARN de interferencia, que silencian genes a través de ARN no codificantes de ∼21pb.
Se postula que una complementariedad total entre el miRNA y el mRNA conduce a la degradación del mRNA, mientras que la complementariedad parcial reprime la traducción proteica. Adicionalmente ciertos miRNA participan en la eliminación de secuencias de ADN y silenciamiento de transposones.
Otra posibilidad es que el simple acto de transcripción de un lncRNA funcione en sí mismo como un mecanismo de acción llamado interferencia transcripcional, donde la transcripción de un gen suprime directamente la transcripción de un segundo gen en cis (es decir, en el mismo alelo), bien sea por competir por el mismo promotor de la transcripción o porque, por ejemplo, el transcrito del lncRNA bloquee el promotor del gen improntado. Además, también se ha observado que deleciones que incluyen a estos lncRNA parecen jugar un papel en la alteración de un correcto establecimiento de la impronta3.
Te invito a profundizar más en este tema echando un ojo a las referencias que te incluyo.
Referencias:

Comentarios
Publicar un comentario